Einsteigen in Python und Programmieren Lernen!
Python hat eine einfache Syntax, es ist einfach in der Anwendung. Ich möchte dir erklären, welche Ziele Python hat und was es braucht, um loszulegen.
Artikel LesenIn diesem Artikel beschäftigen wir uns mit COVID-19, und der Datenanalyse in Python. Die Daten zum Coronavirus sollen uns einen Einblick geben, wie man an die Datenanalyse herangehen kann. Denn zu COVID-19 gibt es Unmengen an Daten, diese aber richtig einzuordnen und zu verstehen ist die Aufgabe, vor der jeder steht.
In diesem Artikel möchte ich dir zeigen wie man mit Python Datenanalyse betreiben kann. Nutze diesen Artikel bitte nicht, um dir klar zu machen, wie es um COVID-19 steht und wie die Entwicklung voranschreitet, ich konnte die Daten nicht zu 100% auf Korrektheit kontrollieren, da ich diese Daten auch nur dem Internet entnommen habe. Um mir sicher zu sein, dass die Daten möglichst richtig sind, habe ich verschiedene Quellen verglichen und geprüft, sodass sie möglichst ähnliche Werte zeigen.
Um den Artikel gut verstehen zu können, solltest du schon etwas Erfahrung mit dem Programmieren haben, da ich nicht nur fertige Datenanalysen von COVID-19-Daten zeigen werde. Am Anfang werde ich dir zeigen wie wir die Daten analysieren können, was wir uns anschauen können und wo wir diese Daten herbekommen.
Python zu können ist übrigens auch ein Vorteil, aber kein Muss. Wenn man andere Programmiersprachen gut kann, sollte man den nachfolgenden Inhalt auch verstehen können, falls Fragen zu meiner Herangehensweise oder meinem Code auftauchen, kannst du mir gerne einen Kommentar schreiben.
Python hat eine einfache Syntax, es ist einfach in der Anwendung. Ich möchte dir erklären, welche Ziele Python hat und was es braucht, um loszulegen.
Artikel LesenEs gibt verschiedene Quellen zu COVID-19-Daten. Inzwischen gibt es auch schon erste API's, welche wir nachfolgend auch nutzen werden, um uns die Daten genauer anzuschauen, denn keiner will die CSV-Daten jeden Tag neu laden. Dafür können wir API's nutzen.
Das bekannteste Daten-Dashboard ist das der Johns Hopkins University. Es ist zu finden unter: https://www.arcgis.com/apps/opsdashboard/index.html#/bda7594740fd40299423467b48e9ecf6.
Ein ähnliches Dashboard gibt es ebenfalls vom Robert Koch-Institut für Deutschland zu finden: https://experience.arcgis.com/experience/478220a4c454480e823b17327b2bf1d4
Wer etwas mehr Hintergrundwissen zum Daten-Dashboard der JHU will, sollte sich den Blogartikel der JHU dazu durchlesen: https://systems.jhu.edu/research/public-health/ncov/
Die JHU stellt ihre gesammelten Daten in einem entsprechenden Github Repository zur Verfügung: https://github.com/CSSEGISandData/COVID-19. Dort findet man unter anderem Daily Reports.
Eine weitere Datenquelle stellt der italienische Zivilschutz als Git Repro zur Verfügung, unter https://github.com/pcm-dpc/COVID-19. Diese Quelle ist auf Italienisch; übersetzt man sich die wichtigen Keywords im JSON, ist diese Datenquelle aber auch gut zu nutzen.
Die OCHA (Amt der Vereinten Nationen für die Koordinierung humanitärer Angelegenheiten) stellt ebenfalls Daten als CSV zur Verfügung, diese Daten entstehen im Zusammenhang mit der Johns Hopkins University. Ebendiese Daten findest du auf der Webseite data.humdata.org.
https://data.humdata.org/dataset/novel-coronavirus-2019-ncov-cases
Die Daten der JHU werden bereits für viele unterschiedliche Projekte genutzt. Eines ist zum Beispiel dieses hier: https://datahub.io/core/covid-19. Die Daten werden dort noch einmal direkt dargestellt, und können in vereinfachter Form direkt abgefragt werden. Dort finden sich auch praktischerweise Anleitungen, wie mit den Daten gearbeitet werden kann.
Der RKI stellt die Daten von seinem Dashboard ebenfalls zur Verfügung: https://npgeo-corona-npgeo-de.hub.arcgis.com/datasetsdd4580c810204019a7b8eb3e0b329dd6_0 Dort finden wir auch demografische Daten wie Altersgruppe und Geschlecht, für genauere Analysen.
Open Data sind Daten, welche meistens von Regierungen zur Verfügung gestellt und für die Allgemeinheit oft über sogenannte Open Data Portale zugänglich gemacht wurden. Wichtig ist, dass die Daten maschinenlesbar sind. Die meisten Daten werden als CSV, JSON oder XML zur Verfügung gestellt. Des Weiteren sollen die Daten kostenlos ohne Bearbeitungsgebühren oder ähnliches, wie bei Open Souce, abrufbar sein.
Ein paar bekannte Portale für Daten sind die folgenden:
Nachdem wir unsere verschiedenen Datenquellen ausfindig gemacht haben, auf Verlässlichkeit geprüft und beschlossen haben, dass wir mit diesen Daten arbeiten werden, können wir uns überlegen wie wir die Daten analysieren wollen bzw. welche Fragestellung wir beantworten wollen.
Als erstes wollen wir eine Verlaufskurve der Daten aus Deutschland nach Datum haben, mit einzelnen Linien für Verstorbene, aktive Fälle, genesene Fälle. Einmal aufgeschlüsselt nach dem Verlauf, also tagesaktuellen Zahlen, und einmal aufbauend auf den Gesamtzahlen.
Als zweites schauen wir uns mithilfe der Daten des RKI das Verhältnis der Geschlechter in Deutschland an, verteilt nach den Landkreisen. Dazu wollen wir die Landkreise, in denen die meisten Frauen und Männer infiziert sind. Danach wollen wir das Ganze auch noch einmal nach Altersgruppen sortieren.
Das jupyter Notebook ist eine Software für interaktive Python-Umgebungen. Dort können wir Code ausführen - die Ausgabe ist direkt darunter. Am Ende, wenn wir mit unserer Analyse fertig sind, können wir dieses Dokument entweder direkt weiterreichen, oder in vielen anderen Formaten zur Verfügung stellen, zum Beispiel in HTML. Diese Variante habe ich genutzt, um euch die Ergebnisse im Folgenden zeigen zu können.
Du kannst nicht nur Cells mit Code anlegen und diese ausführen. Innerhalb deiner Analyse kannst du auch Cells mit Markdown anlegen, um für andere ausführliche Erklärungen zu ergänzen, damit sie mit deinen Daten besser arbeiten bzw. diese verstehen können.
Alternativ könnt ihr euch die jupyter Notebook Dateien, die ich erstellt habe, für diese Analyse direkt runterladen, um euch den Code davon genauer anzuschauen, oder auch weitere Analysen durchzuführen.
Um jupyter Notebooks auszuführen, gibt es viele verschiedene Softwarelösungen
Es gibt verschiedene Module für die Analyse der Daten. Wir benötigen für unsere Datenanalyse 2 Module, die ich euch nun einmal kurz vorstellen möchte.
Die Module werden mittels des PIP Befehles installiert. Mehr Informationen zu PIP findest du im Artikel "Wie du PIP Installieren und Verwenden kannst?".
Um unsere Daten zu analysieren und von unnötigem Ballast zu bereinigen, verwenden wir das Modul Pandas. Dieses Modul bietet die Möglichkeit Daten als CSV oder auch JSON einzulesen. In unseren Beispielen verwenden wir primär CSV-Dateien. Unter anderem können wir auch nach bestimmten Inhalten gruppieren und filtern.
Das Gruppieren werden wir für Analysen vermehrt brauchen. Zum Beispiel, um die Todesfälle nach Altersgruppe oder Bundesländern sortieren zu können. Dafür benötigen wir durch Pandas keine aufwendigen Schleifen, sondern können alles in einer Zeile schnell Gruppieren.
Weitere Funktionen von Pandas sind:
Weitere Informationen zu Pandas gibt es auf der offiziellen Webseite unter: Pandas
Mittels des Moduls matplotlib lassen sich komplexe Liniendiagramme, Kreisdiagramme, Balkendiagramme und viele weitere komplexe Diagramme und Grafiken erstellen. Es arbeitet hervorragend mit dem Numpy Modul und Pandas Modul zusammen. So können wir unsere Daten visualisieren.
So können wir zum Beispiel einen Graphen mit zwei Y-Achsen erstellen um das Verhältnis zwischen Todesfällen und Fällen insgesamt darzustellen, in Bezug auf die Altersgruppe. Danach können wir auch noch die Farben der einzelnen Linien anpassen sowie die Texte der Labels.

Weitere Informationen zu matplotlib gibt es auf der Webseite unter: https://matplotlib.org/
Nachdem wir nun wissen, wo wir unsere Daten herbekommen und womit wir unsere Analyse erstellen können, also mit Python und den Modulen Pandas, Matplotlib und jupyter Notebook, um die Daten in einer gut leserlichen Form abzulegen, können wir mit der eigentlichen Analyse starten.
Als erstes starten wir mit einer Verlaufskurve von Deutschland. Dafür verwenden wir die Daten von https://datahub.io/core/covid-19 - dies sind die Daten der John Hopkins University. Allerdings sind diese schon zu einer CSV zusammengefasst, anstatt wie im Github Repro der JHU. Für die einzelnen Tage wurden die CSV-Dateien zusammengefasst und wir können uns diesen Schritt sparen.
Als erstes importieren wir unsere beiden benötigen Module und definieren für Jupyter Notebooks Matplotlib als inline.
import pandas as pd
import matplotlib.pyplot as plt
%matplotlib inlineDanach laden wir unsere CSV-Datei mit pd.read_csv(). Damit können wir sie mit den Daten aufgeschlüsselt nach Land und Datum lesen. Um zu testen, ob unsere Daten gut aussehen und keine unnötigen Spalten aufweisen, die wir nicht benötigen, holen wir uns mit .head() einmal die ersten 5 Columns.
covid19 = pd.read_csv('countries-aggregated_csv.csv',sep=",")
covid19.head()Nachdem wir dies erledigt haben, müssen wir nur noch unsere Zahlen für die Verlaufskurven vorbereiten. Als erstes müssen wir die Daten filtern, damit wir nur die Daten von Deutschland haben. Das tun wir mit den folgenden Zeilen.
countryData = covid19[covid19["Country"] == "Germany"].copy(deep=True)Dadurch erhalten wir jetzt einen komplett frischen Dataframe mit den Daten von Deutschland.
Wenn wir hier Germany mit irgendeinem anderen Land ersetzen würden, könnten wir uns die Daten für viele andere Länder ausgeben lassen.
Nun erstellen wir uns für Verstorbene, aktuelle Fälle und Genesene drei neue Spalten: DeathsCount, ConfirmedCount und RecoveredCount. Darin speichern wir die Zahl Minus den Wert vom Vortag. Das machen wir mit shift(1); so bekommen wir die Differenz zu den Werten vom Vortag.
for key in ["Deaths","Confirmed","Recovered"]:
countryData.loc[:,(key + "Count")] = countryData[key] - countryData[key].shift(1)Danach erstellen wir noch unsere 2 Plots für die zwei verschiedenen Daten, einmal für die Tageszahlen und einmal für die Zahlen im Verlauf.
plotData = [['Deaths','Confirmed','Recovered'],['DeathsCount','ConfirmedCount','RecoveredCount']]
for key,data in enumerate(plotData):
countryData.reset_index().plot(x ='Date', y=data,color=['black','red','lime'], kind = 'line', stacked=False, figsize=(10,10), linewidth=3)
plt.legend(frameon=True, loc='upper left')
plt.savefig(str(key) + '-data.png',dpi=200,pad_inches=5)Diese machen wir in einer for-Schleife, da, bis auf die Datenfelder, die Plots identisch sein sollen.
Durch die enumerate-Funktion bekommen wir einen Key zu jedem Element in der Liste. So können wir jeden Plot als Grafik mit eindeutigen Namen abspeichern, da ich die Dateien auch noch für den Artikel benötige. Diese könnt ihr zum Beispiel auch nutzen, um später einen Bericht zu erstellen.
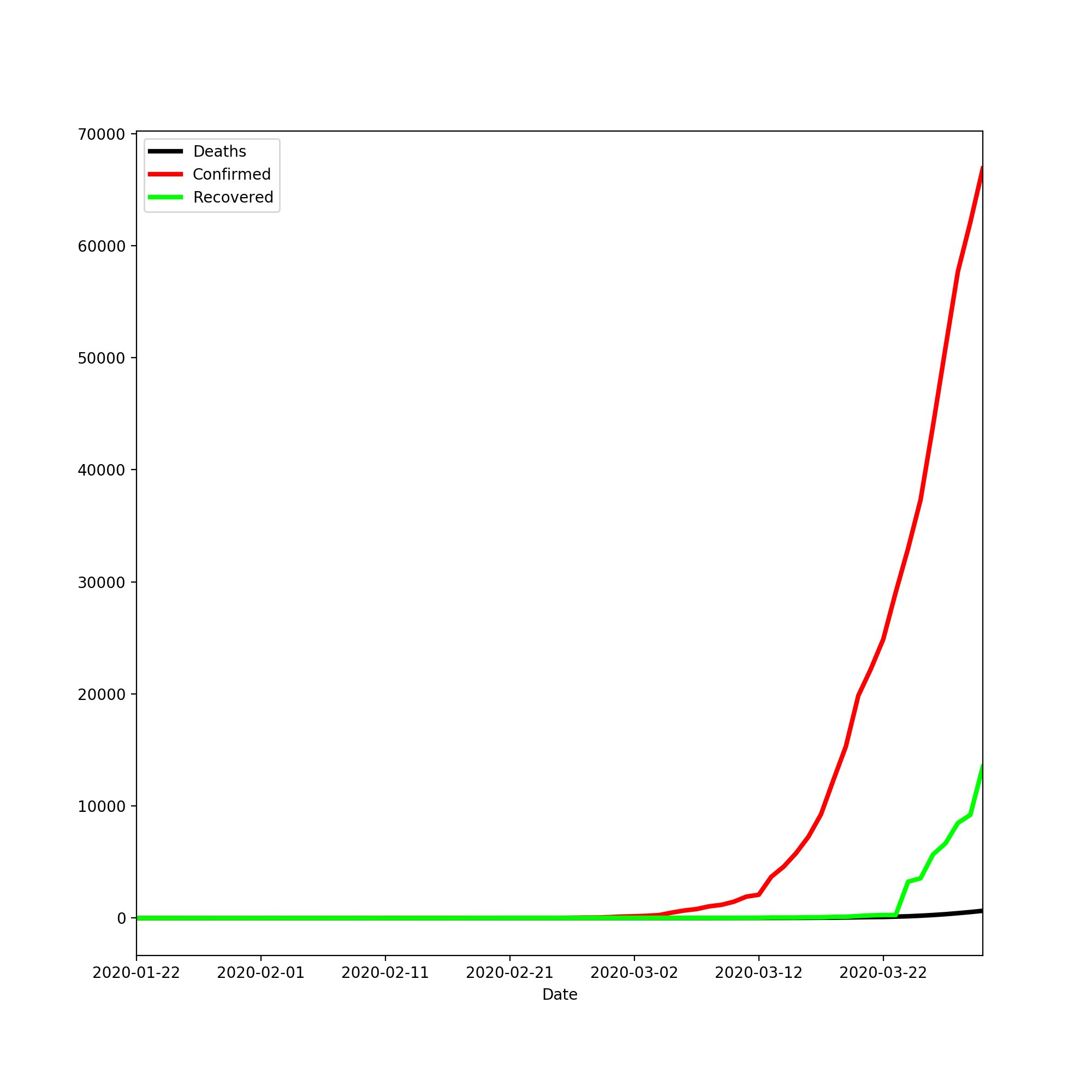
Durch plt.legend platzieren wir die Legende in der oberen linken Ecke. Die fertigen Plots sehen wie folgt aus:

Als nächstes wollen wir die Daten vom Robert Koch Institut für Deutschland genauer analysieren, dafür benötigen wir wieder unsere Imports. Diesmal setzen wir noch einige Optionen, weil unsere Tabellen etwas größer sind, wir uns aber alles ansehen wollen.
import pandas as pd
import matplotlib.pyplot as plt
%matplotlib inline
pd.set_option('display.max_columns', None)
pd.set_option('display.max_rows', None)Dafür benötigen wir die Option, dass wir mehr Rows und Columns sehen können. Um das auf "unlimitiert" zu stellen, benötigen wir die Optionen max_columns und max_rows. Durch None setzen wir diesen Wert auf Unlimited.
RKIData = pd.read_csv('https://opendata.arcgis.com/datasets/dd4580c810204019a7b8eb3e0b329dd6_0.csv');
RKIData.head()Als erstes laden wir unsere Daten mit read_csv, leider konnten wir hier nicht mit der API von https://npgeo-corona-npgeo-de.hub.arcgis.com/dataset/dd4580c810204019a7b8eb3e0b329dd6_0 arbeiten, weil diese nur bis zu 2000 Einträge ausgibt. Die API müsste aber eigentlich 23.000 Einträge enthalten. So haben wir direkt die URL der immer aktuellen CSV eingetragen, um später die Analyse mit aktuelleren Daten wiederholen zu können.
Die Daten vom Robert Koch-Institut in diesem Artikel sind vom 31.03.2020, also Achtung: Falls du die Daten selber analysierst, wirst du andere Ergebnisse erhalten, weil deine Daten aktueller sind.
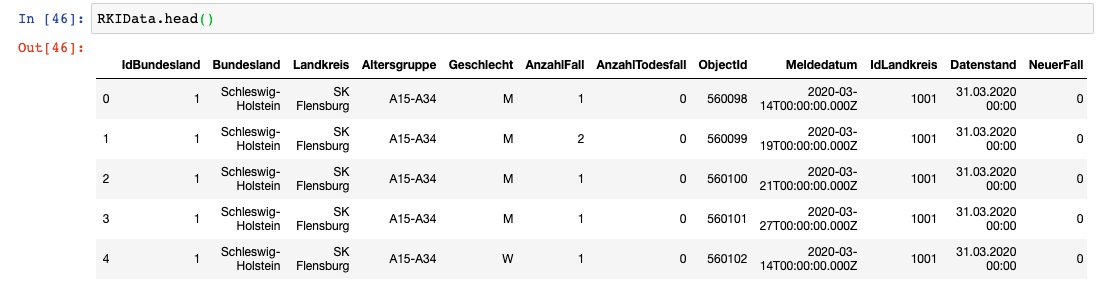
Die ausgegebenen Daten sehen zur Zeit noch aus wie in diesem Screenshot. Dort sind sehr viele Daten enthalten, welche wir gar nicht für die weitere Analyse, die wir machen wollen, benötigen, also bereinigen wir diese. Als erstes löschen wir bestimmte Spalten. Im Anschluss löschen wir Zeilen, die bei Geschlecht oder Altersgruppe einen unbekannten Wert haben, da wir ohne diese Werte arbeiten wollen.
RKIData = RKIData[RKIData.Geschlecht.str.contains("unbekannt") == False]
RKIData = RKIData[RKIData.Altersgruppe.str.contains("unbekannt") == False]
RKIData = RKIData.drop(['NeuerFall','NeuerTodesfall','ObjectId','Meldedatum','IdBundesland','IdLandkreis'],axis=1)
RKIData.head()Die Daten sind nun bereinigt und enthalten nur noch die Werte, für die wir uns interessieren.
Datenstand - 31.03.2020 - Die Daten geben dir einen Überblick darüber wie die Daten vom Robert Koch-Institut aufgebaut sind. Zu den Coronavirus Fällen in Deutschland.
| Bundesland | Landkreis | Altersgruppe | Geschlecht | AnzahlFall | AnzahlTodesfall | Datenstand |
|---|---|---|---|---|---|---|
| Schleswig-Holstein | SK Flensburg | A15-A34 | M | 1 | 0 | 31.03.2020 00:00 |
| Schleswig-Holstein | SK Flensburg | A15-A34 | M | 2 | 0 | 31.03.2020 00:00 |
| Schleswig-Holstein | SK Flensburg | A15-A34 | M | 1 | 0 | 31.03.2020 00:00 |
| Schleswig-Holstein | SK Flensburg | A15-A34 | M | 1 | 0 | 31.03.2020 00:00 |
| Schleswig-Holstein | SK Flensburg | A15-A34 | W | 1 | 0 | 31.03.2020 00:00 |
Nun prüfen wir einmal, ob unsere Daten der Realität entsprechen können. Wir wissen, dass das Coronavirus (COVID-19) in allen deutschen Bundesländern vertreten ist. Also müssen wir in der Spalte Bundesland 16 verschiedene Werte haben. Das können wir mit der folgenden Abfrage überprüfen:
RKIData['Bundesland'].nunique()Die Funktion unique() gibt eine Liste aller Werte in der Spalte Bundesland zurück. Durch das nunique erhalten wir die Länge dieser Liste.
Als nächstes können wir uns noch mit der info()-Methode weitere Informationen zu unserem Datensatz holen. Der Aufruf sieht wie folgt aus:
RKIData.info()Als Ergebnisse erhalten wir die im folgenden Bild gezeigten Informationen. Zum Beispiel finden wir dort die Information, dass wir 25438 Einträge und 7 Spalten in unserer Tabelle haben. Ebenfalls erhalten wir die verschiedenen Datentypen der Spalten.
<class 'pandas.core.frame.DataFrame'>
Int64Index: 25438 entries, 0 to 25775
Data columns (total 7 columns):
Bundesland 25438 non-null object
Landkreis 25438 non-null object
Altersgruppe 25438 non-null object
Geschlecht 25438 non-null object
AnzahlFall 25438 non-null int64
AnzahlTodesfall 25438 non-null int64
Datenstand 25438 non-null object
dtypes: int64(2), object(5)
memory usage: 1.6+ MBStarten wir nun mit der Altersgruppe der Erkrankten in Deutschland. Dafür benötigen wir zwei Methoden, um die Daten zu erhalten. Die groupby Methode und die sum Methode.
Die Groupby Methode gruppiert alle zählbaren Werte bei einer Methode, die man definiert, also Integer und Floats. Die Summen-Methode .sum() summiert die einzelenen Werte, da wir nur zwei Spalten haben. Mit Zahlenwerten werden diese anhand der Altersgruppen gruppiert.
Die Tabelle zeigt die Relation zwischen Fall und Todesfall je nach Altersgruppe.
| Altersgruppe | Anzahl Fall | Anzahl Todesfall |
|---|---|---|
| A00 - A04 | 452 | 0 |
| A05 - A14 | 1203 | 0 |
| A15 - A34 | 15809 | 1 |
| A35 - A59 | 29515 | 30 |
| A60 - A79 | 29515 | 30 |
| A80+ | 3257 | 376 |
Das Ganze nur als Tabelle zu haben wäre langweilig. Deshalb erstellen wir aus dieser Tabelle direkt einen Graphen, um die Werte direkt in einem optischen Verhältnis zu sehen. Dabei arbeiten wir mit zwei Y-Achsen, da die Werte von Todesfall und Gesamtzahl der Fälle eine sehr große Differenz haben. Auf einem Graphen mit einer Y-Achse könnte man dieses nicht mehr gut lesen.
frame = RKIData.groupby(['Altersgruppe']).sum().reset_index()
fig,ax = plt.subplots(figsize=(10,10))
ax.plot(frame['Altersgruppe'], frame['AnzahlTodesfall'], color="black")
ax.set_xlabel("Altersgruppe",fontsize=14)
ax.set_ylabel("Anzahl Todesfall",color="black",fontsize=14)
ax2=ax.twinx()
ax2.plot(frame['Altersgruppe'], frame['AnzahlFall'],color="red")
ax2.set_ylabel("Anzahl Fälle",color="red",fontsize=14)
fig.savefig('data.png',dpi=200,pad_inches=5) # Bild SpeichernAls Ergebnis erhalten wir den folgenden Graphen, mit zwei Linien. Die rote, für die Gesamtzahl der Fälle und die schwarze, für die Todesfälle.
An diesem Graphen können wir zum Beispiel sehen, dass die meisten Fälle zwischen 34 - 59 liegen, aber die meisten Todesfälle bei 80+ in Deutschland liegen.
In diesem Beispiel gruppieren wir wieder. Dieses Mal allerdings am Geschlecht. Wir lassen uns wieder die Summe berechnen. Danach erstellen wir wieder einen Graphen und speichern uns diesen ab. Dieses Mal benutzen wir die Option secondary_y, um unsere Werte in einem besseren Verhältnis darzustellen.
plots = RKIData.groupby(['Geschlecht']).sum().plot(kind="bar",figsize=(20,10),secondary_y="AnzahlTodesfall")
plots.get_figure().savefig('geschlecht.png',dpi=200,pad_inches=5)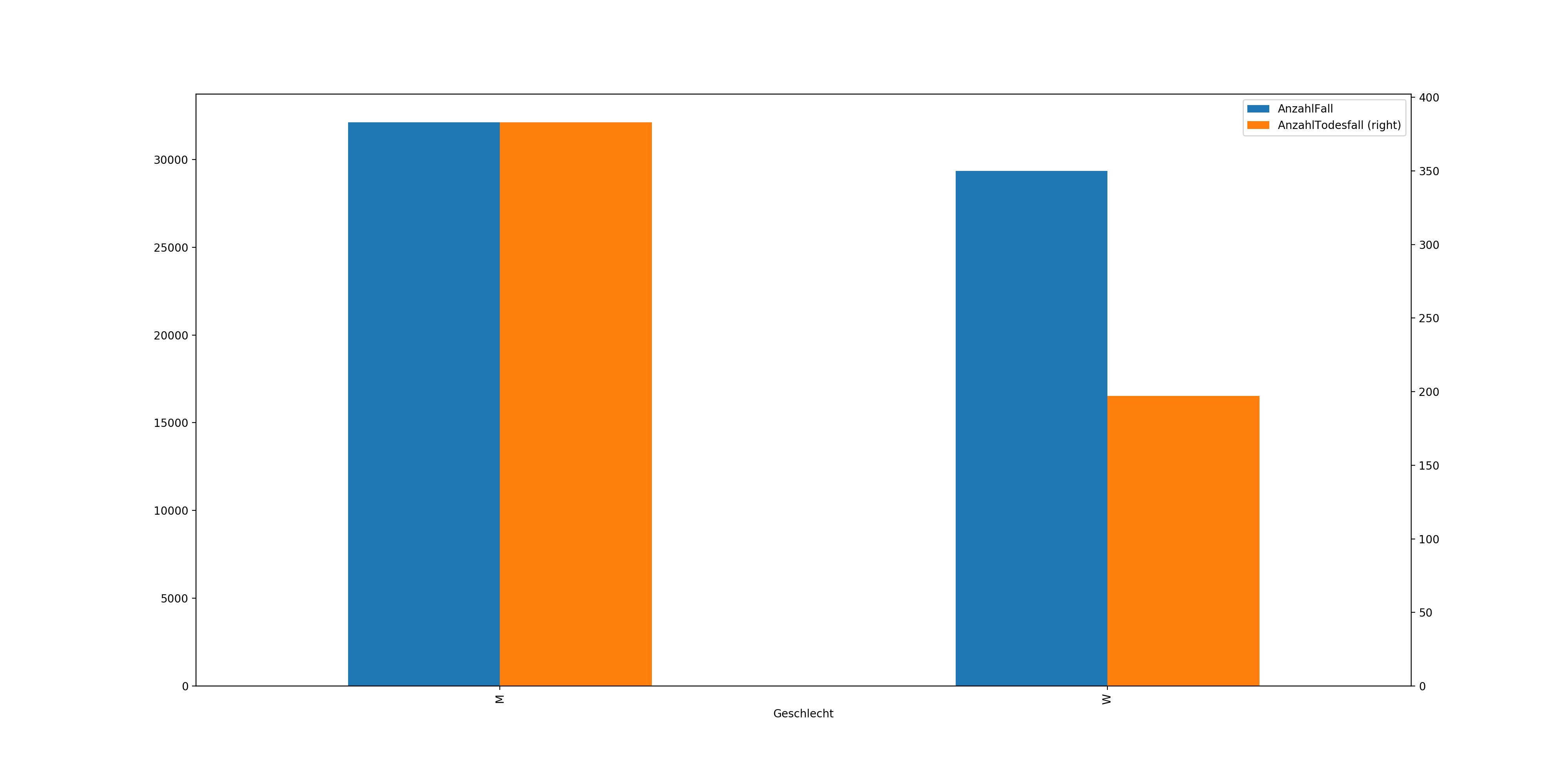
So können wir sehen das Frauen allgemein weniger betroffen sind und diese auch eine deutlich geringere Sterberate haben.
Als nächstes wollen wir nun das Geschlecht und die Altersgruppe vergleichen. Dafür nehmen wir wieder die Groupby-Methode, allerdings mit zwei Parametern. Jetzt sortieren wir unsere Tabelle noch nach der Anzahl der Fälle, da dass für die später Grafik, die wir erstellen wollen, verständlicher ist.
RKIData.groupby(['Geschlecht','Altersgruppe']).sum().sort_values(by=["AnzahlFall"])Als Ergebnisse erhalten wir die folgende Tabelle.
Diese wollen wir nun noch einmal als Bar Plot darstellen, da eine Visualisierung immer ein besseren Eindruck von den Daten verschafft. Wie heißt es so schön: "Ein Bild sagt mehr als tausend Worte".
Diese Grafik zeigt uns, dass dort, wo die meisten Fälle sind, die Todesrate vergleichsweise eher niedrig ist. Was zum Beispiel auch noch auffällt, ist, dass die Todesrate bei Männern im Alter zwischen 35 und 59 deutlich höher ist, obwohl die Fallzahl nur minimal höher ist.
Der fertige Code für diese Grafik sieht wie folgt aus:
GADaten = RKIData.groupby(['Geschlecht','Altersgruppe']).sum().sort_values(by=["AnzahlFall"])
thePlotGADaten = GADaten.plot(kind="bar",secondary_y="AnzahlTodesfall",figsize=(20,10))
thePlotGADaten.get_figure().savefig('geschlecht-altersgruppe.png',dpi=200,pad_inches=5)Nun wollen wir die 10 am stärksten betroffenen Landkreise nach Todes- und Fallzahlen erhalten. Da wir nur die am stärksten betroffenen haben wollen, müssen wir uns die letzten 10 Spalten von der Tabelle holen. Das geht wie folgt.
LandkreisTodesfall = RKIData.groupby(['Landkreis']).sum().sort_values(by=["AnzahlTodesfall"])[-10:].plot(kind="bar",figsize=(20,10))
LandkreisTodesfall.get_figure().savefig('todesfall-landkreis.png',dpi=200,pad_inches=5)Um nur die letzten 10 Einträge zu holen, benötigen wir den Parameter [-10:]. Damit bekommen wir eben nur diese, danach lassen wir wieder unseren Plot erstellen.
Nun können wir sehen, dass, wenn es nach den Todesfällen geht, der Landkreis Heinsberg am stärksten betroffen ist. Nun wechseln wir "AnzahlTodesfall" zu "AnzahlFall" und wir sehen ein komplett anderes Bild.
Dort sehen wir, dass München und Hamburg nach den Fallzahlen deutlich stärker betroffen sind als Heinsberg. Allerdings hat Heinsberg eben die höhere Todesrate im Verhältnis.
Als letztes wollen wir uns noch einmal die am stärksten betroffenen Länder anhand der Todesfälle anschauen. Wir nehmen hierfür wieder die Daten der Quelle https://datahub.io/core/covid-19. Diese haben in der Spalte Deaths, Confirmed, Recovered immer die Gesamtzahl an diesem Tag + die der vergangen Tage. Das heißt, wir brauchen für unsere Analyse nur den letzten Tag. Danach gruppieren wir die Daten nach Ländern und sortieren diese nach Todesfällen. Dann holen wir uns wieder die letzten 10. Diese Analyse ist ähnlich zu der der Landkreisen.
covid19.loc[covid19["Date"] == "2020-03-30"].groupby(['Country']).sum().sort_values("Deaths")[-10:]Als Ergebnisse erhalten wir die folgende Tabelle der am stärksten betroffenen Länder.
Danach erstellen wir uns nun eine Grafik, wo wir die Verstorbenen schwarz einfärben wollen, die Fälle rot und die recovered (genesenen) Fälle sollen grün eingefärbt werden.
plotCountrys = covid19.loc[covid19["Date"] == "2020-03-30"].groupby(['Country']).sum().sort_values("Deaths")[-10:].plot(kind="bar",figsize=(20,10),color=['red','lime','black'])
plotCountrys.get_figure().savefig('countrys.png',dpi=200,pad_inches=5)Danach erstellen wir uns nun eine Grafik, wo wir die Verstorbenen schwarz einfärben wollen, die Fälle rot und die recovered (genesenen) Fälle sollen grün eingefärbt werden.
Eine wichtige Sache solltest du bei der Analyse der internationalen Daten nicht vergessen: Es gibt kein genormtes Messverfahren für die Coronafälle. In Italien wird zum Beispiel auch post mortem ein Test auf Corona durchgeführt, auch, wenn sie nicht direkt an Corona gestorben sind. Diese werden in die Todesstatistik aufgenommen, hingegen wird in Deutschland zum Beispiel kein Test post mortem durchgeführt. Heißt, diese kommen auch nicht in die Statistik. Und so gibt es je nach Land unterschiedliche Feinheiten, weshalb die Daten grundsätzlich immer mit etwas Abstand betrachtet werden sollten.
Als letztes will ich dir noch die 2 Jupyter Notebooks zur Verfügung stellen, mit denen ich gearbeitet habe. Am besten holst du dir die CSV-Dateien einmal neu. Deshalb habe ich diese nicht in die Downloads gestellt. Du findest sie bei den entsprechenden Textabschnitten.
Du fragst dich jetzt, was du noch analysieren kannst? Da gibt es eine Menge! Schau dir doch mal deinen Landkreis, oder dein Bundesland genauer an. Analysiere die Altersgruppen, oder berechne die Todesraten im Verhältnis zu den Fällen. Oder vielleicht findest du ja noch Daten zu den Krankenhausbetten in den verschiedenen Ländern, dann kannst du schauen wie viele Betten im Verhältnis zu den Fällen verfügbar sind.
Hinterlasse mir gerne einen Kommentar zum Artikel und wie er dir weitergeholfen hat beziehungsweise, was dir helfen würde das Thema besser zu verstehen. Oder hast du einen Fehler entdeckt, den ich korrigieren sollte? Schreibe mir auch dazu gerne ein Feedback!
Es sind noch keine Kommentare vorhanden? Sei der/die Erste und verfasse einen Kommentar zum Artikel "Wie Data Science funktioniert - Mit COVID-19 Daten!"!